Il 2024 si apre con buone prospettive nel trattamento delle infezioni batteriche resistenti agli antibiotici e sul futuro della ricerca farmacologica.
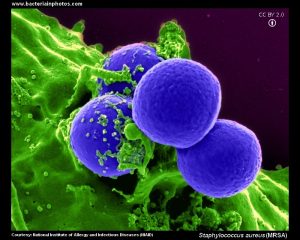
È stata scoperta una nuova classe di antibiotici che, sia in laboratorio che in esperimenti condotti sui topi, si è dimostrata efficace contro i ceppi di Staphylococcus aureus resistenti alla meticillina, i temuti MRSA (Methicillin Resistant Staphylococcus aureus).
Le infezioni causate da ceppi batterici MRSA ogni anno causano oltre 100mila morti in tutto il mondo e, quindi, questa notizia ha suscitato grandi aspettative e speranze. Si consideri, inoltre, che nessun nuovo antibiotico è stato approvato negli ultimi 50 anni contro i batteri Gram-negativi, che presentano pareti cellulari sottili, mentre gli MRSA sono batteri Gram-positivi, che presentano anche pareti cellulari più spesse.
 La scoperta di questa nuova classe di antibiotici è il risultato di uno studio, pubblicato sulla rivista Nature, e si deve ai ricercatori del MIT (Massachusetts Institute of Technology), che hanno utilizzato gli algoritmi dell’Intelligenza Artificiale (AI) e in particolare il “Deep learning” per setacciare circa 12 milioni di composti alla ricerca di nuove molecole.
La scoperta di questa nuova classe di antibiotici è il risultato di uno studio, pubblicato sulla rivista Nature, e si deve ai ricercatori del MIT (Massachusetts Institute of Technology), che hanno utilizzato gli algoritmi dell’Intelligenza Artificiale (AI) e in particolare il “Deep learning” per setacciare circa 12 milioni di composti alla ricerca di nuove molecole.
Un aspetto estremamente interessante è che i ricercatori sono riusciti anche a individuare quali informazioni cercava l’AI e questo potrebbe risultare cruciale per progettare altri farmaci ancora più efficaci di quelli finora identificati. Per raggiungere il loro obiettivo gli scienziati hanno modificato un algoritmo di ricerca già esistente, il “Monte Carlo tree search”, che consente di generare non solo una stima dell’attività antibatterica di ciascuna molecola, ma anche una previsione di quale parte del composto è responsabile di tale proprietà.
Per ridurre ulteriormente i farmaci candidati, i ricercatori hanno affinato la ricerca addestrando ulteriori tre modelli di deep learning per prevedere quali composti fossero tossici per tre diversi tipi di cellule umane, in modo da individuare le molecole che potevano uccidere i microbi con minimi effetti avversi sull’organismo umano. I ricercatori hanno così selezionato circa 280 molecole da testare in laboratorio: due di queste, appartenenti alla stessa classe, sono risultate particolarmente efficaci contro gli MRSA
L’attività sperimentale ha rivelato che i composti sembravano uccidere i batteri alterando la loro capacità di mantenere il Gradiente Protonico Elettrochimico Transmembrana (gpet), un meccanismo vitale fondamentale, attivo nei mitocondri e che permette di generare un potenziale elettrochimico nell’ambito della catena di trasporto degli elettroni. L’energia potenziale dei protoni produce la sintesi finale di ATP, le molecole che le cellule utilizzano per accumulare energia.
Le evidenze sperimentali dimostrano che la nuova classe di molecole può essere attiva contro i patogeni Gram-positivi, dissipando selettivamente la forza motrice protonica nei batteri. Le nuove molecole, inoltre, attaccano selettivamente le membrane cellullari, in modo da non determinare un danno sostanziale alle membrane cellulari umane.
Questo modello di ricerca, basato sull’individuazione di sottostrutture per disegnare composti chimici ex-novo, apre stimolanti prospettive per i ricercatori che potranno anche avvalersi dell’approccio “out of the box”, che ha consentito di individuare gli algoritmi utilizzati dall’AI nel processo di selezione delle nuove molecole.
Leggi anche i nostri articoli sull’antibioticoterapia:
- Nuovi antimicotici in cantiere anche per l’oftalmologia? – Oculista Italiano
- Antibiotici e citotossicità – Oculista Italiano
- Antibiotico resistenza nelle infezioni oculari – Oculista Italiano
- Farmacisti e antibiotico-resistenza – Oculista Italiano
Wong F, Zheng EJ, Valeri JA, Donghia NM, Anahtar MN, Omori S, Li A, Cubillos-Ruiz A, Krishnan A, Jin W, Manson AL, Friedrichs J, Helbig R, Hajian B, Fiejtek DK, Wagner FF, Soutter HH, Earl AM, Stokes JM, Renner LD, Collins JJ. Discovery of a structural class of antibiotics with explainable deep learning. Nature. 2023 Dec 20. doi: 10.1038/s41586-023-06887-8. Epub ahead of print. PMID: 38123686.
